│
└───Data
│ │
│ └───Input
│ │ │
│ │ └───Chronology_setting
│ │ │ │
│ │ │ └───Bchron_crash
│ │ │ │
│ │ │ └───Chron_control_point_types
│ │ │ │
│ │ │ └───Percentage_radiocarbon
│ │ │
│ │ └───Depositional_environment
│ │ │ │
│ │ │ └───Neotoma
│ │ │ │
│ │ │ └───Other
│ │ │
│ │ └───Eco_group
│ │ │
│ │ └───Harmonisation_tables
│ │ │
│ │ └───Neotoma_download
│ │ │
│ │ └───Potential_duplicates
│ │ │
│ │ └───Other
│ │ │
│ │ └───Regional_age_limits
│ │
│ └───Personal_database_storage
│ │
│ └───Processed
│ │
│ └───Chronology
│ │ │
│ │ └───Chron_tables_prepared
│ │ │
│ │ └───Models_full
│ │ │
│ │ └───Predicted_ages
│ │ │
│ │ └───Temporary_output
│ │
│ └───Data_filtered
│ │
│ └───Data_harmonised
│ │
│ └───Data_merged
│ │
│ └───Data_with_chronologies
│ │
│ └───Neotoma_processed
│ │ │
│ │ └───Neotoma_chron_control
│ │ │
│ │ └───Neotoma_dep_env
│ │ │
│ │ └───Neotoma_meta
│ │
│ └───Other
│
└───Outputs
│
└───Data
│
└───Figures
│ │
│ └───Chronology
│ │
│ └───Pollen_diagrams
│
└───Tables
│
└───Meta_and_referencesGuía paso a paso para el procesamiento de datos con FOSSILPOL
Resumen
El flujo de trabajo FOSSILPOL está estructurado de manera modular, donde todos los pasos se organizan secuencialmente y guiados por un único archivo principal de configuración (Config file) donde todos los criterios y configuraciones se predefinen por el usuario.

Figura 1 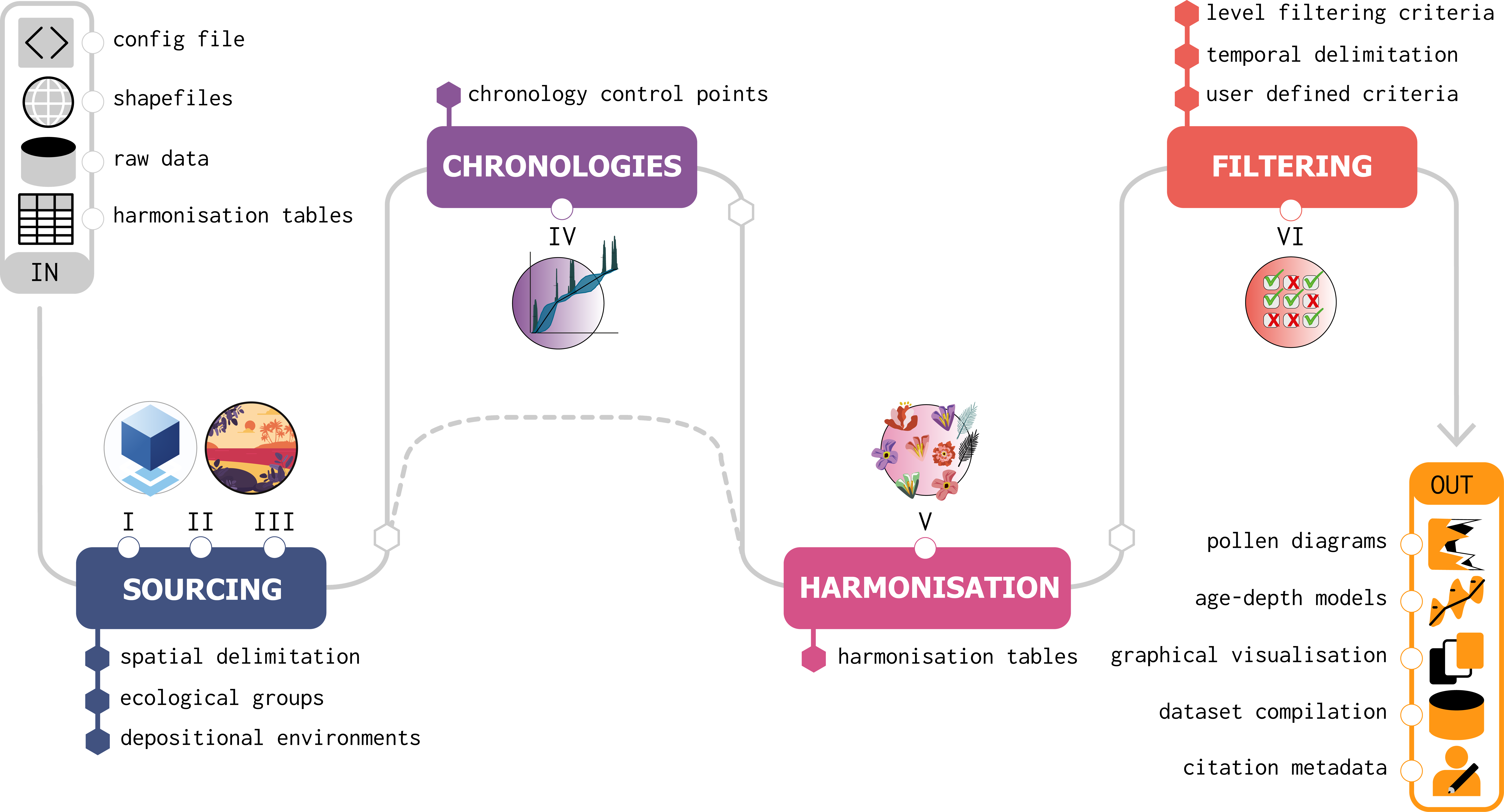
Entrada de datos
El flujo de trabajo FOSSILPOL está configurado de manera que los datos de Neotoma Paleoecological Database (“Neotoma” de aquí en adelante) son la fuente principal de datos. Sin embargo, también se pueden utilizar otras fuentes de datos en paralelo utilizando nuestro formato predefinido (Fig. 2). El usuario, por lo tanto, tiene la flexibilidad de obtener datos de Neotoma o de otra fuente de datos siempre que se utilice nuestro archivo de formato predefinido (ver otras fuentes de datos).
Se requieren tres insumos adicionales para la configuración inicial del flujo de trabajo:
Archivo de configuración (
00_Config_file.R) - contiene todos los ajustes seleccionados por el usuario que se aplicarán a lo largo del flujo de trabajo. Estos van desde configuraciones técnicas (por ejemplo, ubicación del almacenamiento de datos) hasta requisitos específicos (por ejemplo, criterios de filtrado) para los registros que se incluirán. Un resumen de dónde se utilizan los criterios de configuración en el flujo de trabajo se resume en (Fig. 2).Shapefiles geográficos - el flujo de trabajo está configurado internamente para que los datos se procesen por regiones geográficas y los shapefiles se utilizan para asignar la información geográfica relevante a los registros a procesar. Primero, el flujo de trabajo está conceptualizado para un proyecto global, por lo que la estructura general del procesamiento de datos se realiza por continente (es decir,
region= “continent”), pero el usuario puede usar cualquier otra delimitación de interés. El flujo de trabajo viene con un shapefile por defecto que delimita aproximadamente los continentes, pero puede ajustarse o reemplazarse según las necesidades del proyecto. En segundo lugar, la armonización taxonómica de los registros se estructura por regiones de armonización proporcionadas por elharmonisation region shapefile. Por defecto, este shapefile es una copia del shapefile continental, pero como las tablas de armonización son específicas por región (ver siguiente ítem de insumo), este shapefile debe ajustarse para representar la delimitación geográfica de las regiones de armonización utilizadas. Finalmente, si el usuario está interesado en otras unidades biogeográficas, climáticas o ecológicas de interés para ser vinculadas a cada registro (por ejemplo, ecorregiones, tipo de bioma, zonas climáticas), entonces se pueden agregar shapefiles (o archivos TIF) adicionales al flujo de trabajo (ver detalles aquí).Tablas de armonización - en cada proyecto, se debe proporcionar una tabla de armonización por región de armonización (delimitada por el correspondiente shapefile de región de armonización, ver arriba). Una tabla de armonización siempre viene con dos columnas: i)
original taxa(taxones originales) con los nombres taxonómicos presentes originalmente en Neotoma y/o en otra fuente de datos en el proyecto, y ii)level_1(taxones armonizados) con los nombres taxonómicos estandarizados. El flujo de trabajo detectará si el usuario ha proporcionado una tabla de armonización o, de lo contrario, creará una nueva tabla con todos los nombres de taxones brutos detectados para cada región de armonización. Esta última puede servir como plantilla para la armonización en la columnalevel_1(ver detalles aquí).
Figura 2 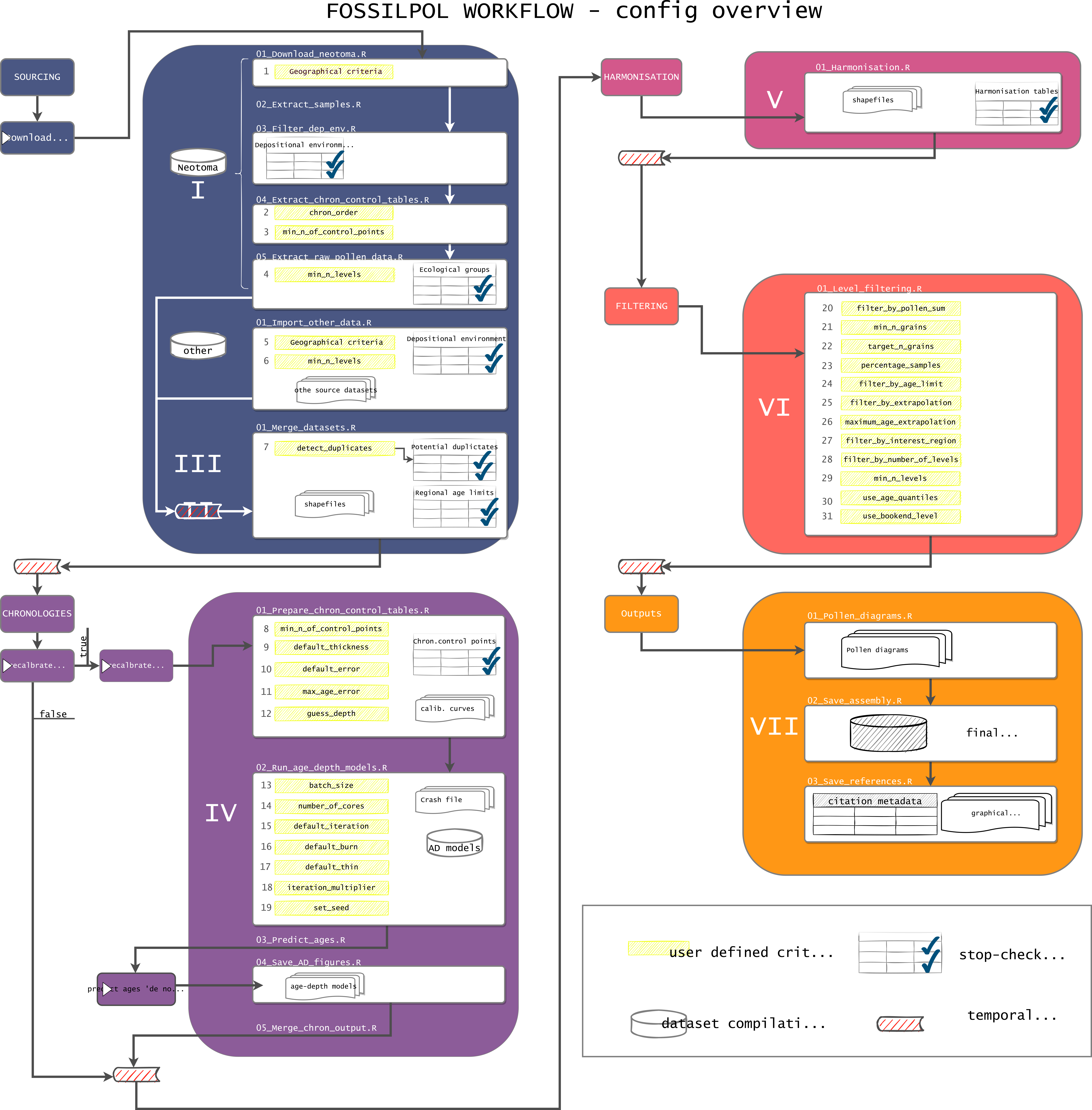
Almacenamiento de datos
El flujo de trabajo producirá varios archivos, incluyendo archivos de salida temporales, tablas de stop-check, y salidas finales (compilación de datos, figuras, etc.):
Archivos temporales de salida: el flujo de trabajo está configurado para que los archivos de datos temporales (en proceso) se guarden en varias etapas del flujo de trabajo. Cada archivo contendrá la fecha de creación para una organización más fácil. Cuando se ejecuta varias veces, el flujo de trabajo detectará automáticamente si hay cambios en un archivo seleccionado y solo lo sobrescribirá si se produce un archivo actualizado (esto lo gestiona el paquete {RUtilpol}). Esto también significa que el usuario no tiene que volver a ejecutar todo el flujo de trabajo, sino que puede volver a ejecutar solo partes específicas. Como el tamaño total de los archivos puede ser considerable, el usuario puede especificar si todos los archivos deben almacenarse dentro de la carpeta del proyecto (por defecto) o en otro directorio (especificado usando
data_storage_pathen el archivo de configuración). Con tal especificación, y después de ejecutar el script00_Config_file.R, se creará una estructura de carpetas adicional (ver [FLAG]).Tablas CSV de stop-check: al ejecutar el flujo de trabajo, habrá varias ocasiones en que se pedirá al usuario que revise y, cuando sea necesario, ajuste las tablas CSV producidas para continuar con el flujo de trabajo (es decir, volver a ejecutar el script). Esto se hace para obligar al usuario a comprobar los resultados intermedios antes de continuar. Por ejemplo, en cierto punto, el flujo de trabajo producirá una lista de todos los grupos ecológicos detectados en la compilación de datos obtenida de Neotoma. Luego, el usuario debe editar la tabla CSV mencionada y especificar qué grupos ecológicos deben mantenerse (
include=TRUE) y cuáles deben filtrarse (include=FALSE). Ten en cuenta que hay varios stop-checks a lo largo del flujo de trabajo (ver resumen en Fig. 2).Salida del flujo de trabajo (
Outputs/, ver Sección VII para más información):- una compilación de datos de polen fósil armonizada taxonómicamente y estandarizada temporalmente, lista para el análisis (formato rds)
- gráficos de curvas de edad-profundidad modeladas para cada registro (formato pdf)
- diagramas de polen de cada registro (formato pdf)
- tabla de metadatos con el principal contribuyente de datos, información de contacto y publicaciones correspondientes para propósitos de citación de los conjuntos de datos utilizados (PDF).
- reproducibility bundle, un archivo zip que contiene todas las secciones importantes para la reproducibilidad de todo el proyecto.
- figuras resumen de la distribución espacial y temporal de la compilación de datos, es decir, un mapa y un gráfico de la duración de los registros, respectivamente (PDF).
Bloque de código 3
Procesamiento de datos
Aquí nos centramos en los scripts dentro de la carpeta R/01_Data_processing que representan todos los pasos necesarios para el procesamiento de datos (desde la obtención de los datos hasta la compilación final del conjunto de datos), organizados en las siguientes Secciones:
Obtención de datos:
/01_Neotoma_source/- obtener y procesar datos de NeotomaObtención de datos:
/02_Other_source/- procesar datos de otras fuentes (opcional)Procesamiento de datos inicial:
/03_Merging_and_geographic_delineation/- combinar fuentes de datos, filtrar duplicados y asignar valores según la ubicación geográficaCronologías:
/04_Chronologies/- preparar tablas de control de cronología, calcular modelos edad-profundidad y predecir edades para los nivelesArmonización:
/05_Harmonisation/- preparar todas las tablas de armonización y armonizar los taxones de polen (morfotipos)Filtrado de datos:
/06_Main_filtering/- filtrar niveles y registros según criterios definidos por el usuarioSalidas:
/07_Outputs/- guardar la salida final incluyendo la compilación de datos, diagramas de polen, información de metadatos, resumen gráfico y reproducibility bundle
I. Obtención de datos: /01_Neotoma_source/
Run_01_01.R- ejecuta todos los scripts dentro de esta carpeta01_Download_neotoma.R- descarga los datos de polen desde la base de datos Neotoma02_Extract_samples.R- crea una tabla a partir de las listas descargadas de Neotoma y descarga la información de los autores03_Filter_dep_env.R- obtiene los datos del ambiente de depósito y filtra registros según las preferencias del usuario04_Extract_chron_control_tables.R- obtiene las cronologías, incluyendo la tabla preferida con los puntos de control de cronología05_Extract_raw_pollen_data.R- extrae los conteos de polen brutos de Neotoma y filtra por grupos ecológicos seleccionados por el usuario.
01_Download_neotoma.R
Todos los registros de polen se descargan de Neotoma en base a los criterios geográficos (extensión espacial, Fig. 2 - criterio config 1) y el tipo de dato seleccionado, en este caso: “pollen”. Observa que se puede utilizar una extensión espacial más compleja, como un polígono, con el argumento loc en RFossilpol::proc_neo_get_all_neotoma_datasets() (ver uso de loc en el ejemplo de neotoma2 aquí).
02_Extract_samples.R
Cada registro se procesa utilizando un ID único de dataset (dataset_id) con la información de metadatos extraída. Los metadatos incluyen información sobre el nombre del registro, información geográfica y los autores y DOI de la publicación conectada al dataset. Los autores y su vínculo con el dataset se guardan en una base de datos de Autor-Dataset creada específicamente para cada proyecto. Esto permite la fácil extracción de autores y DOI para la compilación final producida por el flujo de trabajo.
03_Filter_dep_env.R
La información de depósito de cada registro ofrece información sobre los ambientes donde se extrajo el registro. Según la pregunta de investigación, puede haber preferencia por ciertos ambientes (por ejemplo, terrestres vs. marinos). Actualmente en Neotoma, los datos sobre ambientes de depósito están organizados en una estructura jerárquica (por ejemplo, “Pond” está anidado en “Natural Lake”, que a su vez está anidado en “Lacustrine”), donde el número máximo de capas anidadas es cinco. En el nivel jerárquico más bajo, actualmente existen más de 50 categorías diferentes de ambientes de depósito (para registros de polen fósil). Según los registros seleccionados, el flujo de trabajo producirá una lista de todos los ambientes de depósito (y su posición jerárquica) presentes en la selección de datos del usuario. Luego se solicita al usuario que defina los ambientes de elección (esto es un punto stop-check, Fig. 2). Observa que excluir ambientes de depósito con una posición jerárquica superior no excluye automáticamente todos los ambientes de depósito anidados en él.
04_Extract_chron_control_tables.R
Los datos de cronología para cada registro están disponibles en una tabla que contiene información sobre los puntos de control de cronología usados para construir un modelo edad-profundidad. Algunos registros pueden tener múltiples tablas de cronología, ya que han sido utilizados para varios proyectos o recalibrados por los responsables de los datos. Estas tablas se enumeran según el orden en que se crearon y subieron. Cada cronología viene con la unidad de edad de la salida del modelo edad-profundidad (por ejemplo, “Radiocarbon years BP”, “Calibrated radiocarbon years BP”) y el rango temporal del registro (edad más joven y más vieja). Las cronologías en “Radiocarbon years BP” suelen ser cronologías más antiguas ya que es práctica común recalibrar el material datado por radiocarbono y producir cronologías expresadas en “Calibrated radiocarbon years BP”. Nota: Las cronologías en “Calibrated radiocarbon years BP” aún vienen con tablas de cronología que contienen las edades de radiocarbono sin calibrar y deben ser calibradas por el usuario si se desea un nuevo modelo edad-profundidad. El flujo de trabajo selecciona automáticamente una tabla por registro según el orden definido por chron_order en el archivo de configuración (Fig. 2 - criterio config 2). Nota: si hay varias tablas con el mismo tipo de unidad de edad (por ejemplo, Calibrated radiocarbon years BP), el flujo de trabajo optará por la tabla más reciente. El usuario puede especificar su preferencia para ciertos tipos de unidad de edad en el archivo de configuración. Además, solo se utilizarán los registros que tengan al menos un número mínimo de puntos de control (definido por min_n_of_control_points en el archivo de configuración, Fig. 2 - criterio config 3).
05_Extract_raw_pollen_data.R
Cada nivel de cada registro incluye información adicional: a) ID único de muestra (sample_id), b) información sobre la profundidad (y edad estimada posteriormente), y c) conteos de polen para cada taxón presente en ese nivel. La información sobre los niveles se divide en dos tablas diferentes (primero con profundidad y edades, y segundo con conteos de polen) vinculadas por sample_id (sample_id).
El flujo de trabajo solo mantendrá los registros con un número mínimo de niveles según lo definido en el archivo de configuración (min_n_levels, Fig. 2 - criterio config 4). El número mínimo de niveles por defecto es tres, pero el usuario puede cambiar este ajuste.
En el caso de los datos obtenidos de Neotoma, cada taxón de polen tiene información sobre el grupo ecológico (por ejemplo, palmas, manglares, etc.). Según los registros seleccionados, el flujo de trabajo producirá una lista completa de todos los grupos ecológicos tras lo cual se solicita al usuario definir qué grupos ecológicos incluir (un punto stop-check, Fig. 2, ver explicación de la abreviatura en Tabla 1).
Tabla 1. Grupos ecológicos asignados a taxones de polen según Neotoma
| ABREVIATURA | GRUPO ECOLÓGICO |
|---|---|
| ACRI | Acritarcos |
| ANAC | Anacrónicos |
| ALGA | Algas (ej. Botryococcus) |
| AQB | Acuáticos (ej. Sphagnum) |
| AQVP | Plantas vasculares acuáticas (ej. Isoetes) |
| BIOM | Mediciones biométricas |
| EMBR | Embriófitas |
| FUNG | Hongos |
| LABO | Análisis de laboratorio |
| MAN | Manglares |
| PALM | Palmas |
| PLNT | Planta |
| SEED | No identificado, pero definitivamente polen - rango o clado de espermatófitos |
| SUCC | Suculentas |
| TRSH | Árboles y arbustos |
| UNID | Desconocido e indeterminable |
| UPBR | Briófitas de tierras altas |
| UPHE | Hierbas de tierras altas |
| VACR | Criptógamas vasculares terrestres |
| VASC | Plantas vasculares |
II. Obtención de datos: /02_Other_source/
Run_01_02.R- ejecuta todos los scripts dentro de esta carpeta01_Import_other_data.R- obtener otras fuentes de datos y filtrar los registros de manera similar a Neotoma.
Nota sobre otras fuentes de datos
Nuestro flujo de trabajo FOSSILPOL permite el uso de otras fuentes de datos en combinación con los datos de Neotoma. Incluir otras fuentes es completamente opcional y puede omitirse según lo indicado por use_other_datasource = TRUE/FALSE en el archivo de configuración.
Se pueden usar cualquier tipo de datos, siempre y cuando contengan la siguiente información obligatoria: a) metadatos, b) ambiente de depósito, c) cronología, d) nivel (edad-profundidad), y e) conteos de polen. Para preparar los datos para su uso, el usuario debe descargar la plantilla de archivo especialmente preparada para este fin. Cada registro de polen debe guardarse como un archivo separado con un nombre único. Se recomienda, por ejemplo, private_data_(nombre_del_sitio).xlsx. El nombre del sitio en el nombre del archivo es crucial ya que se comparará con todos los demás registros de polen en Neotoma para detectar posibles duplicados en una etapa posterior del flujo de trabajo. Todos los archivos deben almacenarse en /Data/Input/Other/ (o según lo especificado por el argumento dir_files, ver abajo).
01_Import_other_data.R
La obtención de otras fuentes de datos sigue un orden simple de acciones:
Los archivos de datos deben ser preparados por el usuario siguiendo la plantilla, un registro por archivo.
Los datos se extraen y formatean para ser compatibles con los datos de Neotoma utilizando la función
RFossilpol::import_datasets_from_folder(), con los siguientes argumentos:dir_files- el usuario puede especificar qué carpeta contiene los datos preparados (por defecto =Data/Input/Other/)suffix- argumento para identificar la fuente de los datos. Por defecto se define como"other", lo que significa que los conjuntos de datos se pueden identificar fácilmente ya que su nombre será(dataset id)_othersource_of_data- marcará la fuente de cada conjunto de datos en la compilación en el resumen de metadatos (ver sección VII). Por defecto se define como"personal_data"data_publicity- marcará la publicidad de los datos de cada conjunto en la compilación en el resumen de metadatos (ver sección VII - Outputs). Por defecto se define como"restricted"pollen_percentage- los conteos de polen medidos como proporciones (por ejemplo, escaneados de diagramas de polen) se pueden marcar aquí. Por defecto enFALSE
Los nombres de los contribuyentes de datos se extraen y se añaden a la base de datos Autor-Dataset usada para la atribución de autor-dataset (ver sección VII - Outputs).
Los datos se tratan de manera similar a los datos de Neotoma, en cuanto al filtrado por ubicación geográfica, número de niveles (Fig. 2 - criterios config 5, 6), y ambientes de depósito (punto stop-check, Fig. 2).
III. Procesamiento de datos inicial: /03_Merging_and_geographic_delineation/
Run_01_03.R- ejecuta todos los scripts dentro de esta carpeta01_Merge_datasets.R- combinar datos de todas las fuentes, filtrar duplicados y asignar valores según la ubicación geográfica
01_Merge_datasets.R
Después del procesamiento inicial, los registros de Neotoma y otras fuentes se combinan.
Detección de duplicados
Existe la posibilidad de que algunos conjuntos de datos de otras fuentes ya estén en Neotoma. Para evitar duplicados en la compilación final, el flujo de trabajo comparará conjuntos de ambas fuentes e identificará posibles duplicados. Este paso es opcional, pero se recomienda seguirlo. Para ello, el usuario debe especificar detect_duplicates == TRUE en el archivo de configuración (esto es predeterminado, Fig. 2 - criterio config 7). El flujo de trabajo iniciará una subrutina simple que utiliza la función RFossilpol::proc_filter_out_duplicates(). Dado que comparar todos los registros de cada fuente de datos entre sí es relativamente exigente en términos de cálculo, la función dividirá los datos en varios grupos según su ubicación geográfica (aprox. 100 registros por grupo). El usuario puede definir el número de grupos mediante el argumento n_subgroups. A continuación, cada registro de una fuente se compara con todos los registros de la otra fuente, siempre que se encuentren en un radio de 1 grado (se asume que los registros duplicados estarán en una ubicación similar). El usuario puede definir la distancia máxima mediante el argumento max_degree_distance. Finalmente, el flujo de trabajo generará una lista de posibles registros duplicados (un punto de stop-check, Fig. 2). Para cada par de registros, el usuario debe especificar qué registros deben eliminarse escribiendo 1 (eliminar el Neotoma) o 2 (eliminar la otra fuente de datos) en la columna delete de la lista creada (dejar 0 dejará ambos registros).
Preparación adicional de datos
Se realizan varios pasos adicionales para crear la compilación completamente combinada antes de pasar al paso de cronología (no requieren acción del usuario):
Todos los nombres de taxones se transforman en un formato más fácil de manejar por ordenador para su manipulación. La función
RFossilpol::proc_clean_count_names()primero transforma los caracteres especiales a texto (por ejemplo,+a_plus_) y luego utiliza {janitor} para transformar en el estilo “snake_case”. Además, el usuario puede especificar cambios adicionales mediante el argumentouser_name_patterns(ver ejemplo en el script). Durante la limpieza de nombres de taxones, el flujo de trabajo guardará lataxa_reference_tablepara la trazabilidad a la taxonomía de Neotoma. Lataxa_reference_tablees un archivo CSV que se guarda en la misma carpeta que las tablas de armonización (Data/Input/Harmonisation_tables/). Más información sobre el proceso de armonización.Los niveles individuales (profundidades de muestra) se ordenan por su profundidad para cada registro con
RFossilpol::proc_prepare_raw_count_levels(). Esto incluye subrutinas, por ejemplo, mantener solo los niveles presentes en todas las tablas de datos, filtrar niveles sin datos de polen y taxones que no estén presentes en ningún nivel.Se asigna información espacial a cada registro según los shapefiles geográficos proporcionados. Específicamente:
Información de región - el shapefile en
Data/Input/Spatial/Regions_shapefileasignará los nombres regionales a cada registro (ver Sección Entrada de datos). El usuario puede (y se recomienda) cambiar la delimitación espacial alterando o reemplazando el shapefile.Delimitación política (países) - obtenido de la base de datos GADM, versión 2.8, noviembre 2015.
Región de armonización - el shapefile en
Data/Input/Spatial/Harmonisation_regions_shapefileasignará la región de armonización (para vincular la tabla de armonización correspondiente; ver Sección Entrada de datos). El shapefile predeterminado en el flujo de trabajo es una copia del shapefile de información regional, pero debe ajustarse para corresponder al área cubierta por las distintas tablas de armonización.Curvas de calibración (normal y post-bomba) - dependiendo de la posición geográfica del registro, se debe asignar una curva de calibración distinta, ya que se usan curvas diferentes para los hemisferios norte y sur, y para ambientes terrestres y marinos. Ver más detalles sobre curvas de calibración.
Adicional - El usuario puede añadir cualquier otra delimitación espacial (por ejemplo, ecozonas). Esto requerirá agregar el shapefile (o archivo TIF) específico en
/Data/Input/Spatial/NOMBRE_DE_LA_CARPETAy ajustar el código R manualmente (optional_info_to_assign) para que el shapefile se lea y su información se asigne a cada registro (ver el ejemplo en el script).
El flujo de trabajo creará una nueva tabla con los límites de edad para cada región representada en los datos, que debe ser editada por el usuario (un punto stop-check, Fig. 2). Por ejemplo, la tabla
Regional_age_limitstendrá los siguientes valores:young_age= edad más joven que debe tener el registroold_age= edad más antigua que debe tener el registroend_of_interest_period= niveles más allá de esta edad serán omitidos
IV. Cronologías: /04_Chronologies/
Run_01_04.R- ejecuta todos los scripts dentro de esta carpeta01_Prepare_chron_control_tables.R- prepara las tablas de cronología para el modelado edad-profundidad02_Run_age_depth_models.R- crea modelos edad-profundidad con BChron03_Predict_ages.R- estima las edades de los niveles individuales04_Save_AD_figures.R- guarda la salida visual de los modelos edad-profundidad en formato pdf05_Merge_chron_output.R- enlaza los modelos edad-profundidad a los conjuntos de datos correspondientes
Nota sobre el modelado edad-profundidad
Para estimar la edad de los niveles individuales en función de su profundidad, se debe construir un modelo edad-profundidad basado en los datos de cronología del registro. Un modelo edad-profundidad proporciona estimaciones de edad para cada nivel y el rango completo de edades del registro.
El modelado edad-profundidad puede ser muy computacionalmente intensivo y llevar bastante tiempo. Por ello, el flujo de trabajo procesa automáticamente varios archivos (formato rds):
- Tablas de control de cronología:
/Data/Processed/Chronology/Chron_tables_prepared/chron_tables_prepared*.rdscontiene todas las tablas de control de cronología preparadas para recalibrarse
- Modelos edad-profundidad:
/Data/Processed/Chronology/Models_full/*contiene un archivo para cada modelo edad-profundidad
- Edades predichas:
/Data/Processed/Chronology/Predicted_ages/predicted_ages*.rdscontiene las edades predichas usando los modelos edad-profundidad
Puede ser no preferible volver a ejecutar todos los modelos edad-profundidad cada vez que se quiera volver a correr el flujo de trabajo. Por eso, el flujo permite guardar los modelos exitosos (Modelos edad-profundidad y Edades predichas) y mantenerlos entre ejecuciones. Esto se puede hacer de varias formas:
recalib_AD_modelsen el archivo de configuración puede fijarse enFALSE. Así se omiten completamente los scripts destinados a calcular los modelos edad-profundidad, y este paso se saltará. Esto solo funciona si los modelos ya fueron creados exitosamente al menos una vez (Edades predichas existen).calc_AD_models_denovoen el archivo de configuración puede fijarse enFALSE(es el valor predeterminado). Si está enTRUE, el flujo no usará los archivos de Modelos edad-profundidad y recalibrará todo “de novo”.predict_ages_denovoen el archivo de configuración está enFALSEpor defecto. Si está enTRUE, el flujo usará todos los archivos de modelos edad-profundidad pero reasignará las edades a cada nivel de todos los registros relacionados (incluso si ya se predijeron exitosamente antes). Esto es útil, por ejemplo, cuando el número de niveles en un registro aumenta desde la última ejecución y esos niveles aún necesitan edades.
IMPORTANTE: Si seleccionas ambos calc_AD_models_denovo y predict_ages_denovo como FALSE en tu primera ejecución de modelado edad-profundidad, el flujo puede pedirte temporalmente cambiarlos a TRUE en la consola (no tienes que modificar el archivo de configuración).
01_Prepare_chron_control_tables.R
Los modelos edad-profundidad se construyen usando puntos de control de cronología (normalmente fechas de radiocarbono) con profundidad conocida, edad estimada y su incertidumbre asociada. Cada registro puede y debe tener varios puntos guardados en la tabla de control de cronología. Cada punto de control tiene las siguientes propiedades:
- Profundidad
- Edad estimada
- Error de la edad estimada
- Tipo de punto de control de cronología
- Curva de calibración utilizada (necesaria para convertir edades de radiocarbono a edades de calendario; ¡ojo! las edades de radiocarbono no son edades de calendario verdaderas).
Este script realiza varios pasos para preparar los registros para el modelado edad-profundidad:
- Crear y adjuntar las curvas de calibración necesarias
- Seleccionar los tipos preferidos de puntos de control de cronología
- Preparar tablas de cronología (incluido corregir problemas con porcentaje de carbono si es necesario)
Curvas de calibración
Las curvas de calibración se asignan a cada punto de control según varios criterios. Si un punto de control tiene un tipo marcado para calibrar (ver sección siguiente), la curva se asigna según la posición geográfica del registro. Solo los puntos de control con edades de radiocarbono sin calibrar requieren recalibración. FOSSILPOL incluye un shapefile basado en la figura 7 de Hogg et al (2020) para asignar correctamente IntCal20, SHCal20 y una curva mixta. Se sigue la recomendación: “…el uso de (i) IntCal20 para áreas al norte de la ITCZ en junio-agosto (líneas discontinuas en la Figura 7) que reciben masas de aire del hemisferio norte durante todo el año, (ii) SHCal20 para áreas al sur de la ITCZ en diciembre-febrero (líneas punteadas en la Figura 7) que reciben masas de aire del hemisferio sur durante todo el año, y (iii) una curva mixta para áreas entre las dos posiciones estacionales de la ITCZ mostradas en la Figura 7, que reciben masas de aire del norte en diciembre-febrero y masas de aire del sur en junio-agosto”. FOSSILPOL también incluye una curva mixta construida con el paquete {rcarbon} con una proporción de contribución de la curva de 1:1. Consulte más información sobre el uso de la curva de calibración mixta.
Todas las curvas de calibración tienen un límite de edad, es decir, solo pueden usarse para ciertas edades. Las curvas de calibración de radiocarbono actualmente no cubren edades mayores a 55 mil años. Para edades más jóvenes, durante el último siglo, existen problemas con el radiocarbono atmosférico y debe usarse un conjunto distinto de curvas (debido a las detonaciones nucleares en los 50-60, que alteraron la señal atmosférica). Si el punto de control tiene una edad de radiocarbono menor a 200 años BP, se usa una curva post-bomba (ver más arriba). Al igual que las curvas normales, la posición geográfica determina qué curva post-bomba asignar.
Las fechas modernas de radiocarbono se calibran usando una de las curvas post-bomba (nh_zone_1, nh_zone_2, nh_zone_3, sh_zone_1_2, sh_zone_3), siguiendo a Hua et al., 2013 y enlace. El flujo asignará la curva adecuada automáticamente (ver función IntCal::copyCalibrationCurve()). Si detecta fechas modernas, mostrará los registros y aplicará la conversión usando el paquete {IntCal}.
Tipos de puntos de control de cronología
Cada punto de control tiene varias propiedades: ID único, profundidad, edad, error, espesor y tipo de control (ej. radiocarbono, biostratigráfico, laminaciones anuales, tefra). Cada tipo tiene diferentes incertidumbres de edad. Por ejemplo, muchos registros antiguos se basan en técnicas indirectas (biostratigráficas, tefra, etc.) y pueden tener grandes incertidumbres. Neotoma tiene más de 50 tipos distintos de puntos de control, que abarcan geocronología (ej. plomo-210, radiocarbono, uranio), escala temporal relativa (por ejemplo, MIS5e, Heinrich Stadial 1, Late Wisconsin event), estratigráfica (por ejemplo, eventos bioestratigráficos como la introducción de taxones antropogénicos), cultural (por ejemplo, European Settlement Horizon), otros métodos de datación absoluta (por ejemplo, laminaciones anuales, fecha de recolección) y otros métodos de datación (por ejemplo, extrapolación o conjeturas). Solo los puntos de control cronológico en edades radiocarbónicas sin calibrar requieren recalibración con las curvas de calibración, ya que la mayoría, si no todos, los demás puntos de control estarán en edades calendáricas y no se debe implementar ninguna recalibración.
El usuario puede elegir qué tipos de control aceptar “tal cual” y cuáles calibrar (un punto stop-check, Fig. 2). El flujo genera automáticamente una lista con todos los puntos de control detectados, incluyendo columnas include y calibrate. En la primera, el usuario indica si se debe incluir el punto; en la segunda, si debe calibrarse.
Preparación de las tablas de cronología
Las tablas de control de cronología deben pasar varios ajustes definidos por el usuario:
- filtrar tipos de puntos de control no deseados seleccionados por el usuario (stop-check)
- filtrar registros que no cumplan el número mínimo de puntos de control (
min_n_of_control_pointsen el archivo de configuración, por defecto =2, Fig. 2 - criterio config 8) - corregir valores ausentes con
default_thickness(por defecto =1, Fig. 2 - criterio config 9) ydefault_error(por defecto =100, Fig. 2 - criterio config 10) - filtrar puntos de control con errores demasiado grandes (
max_age_erroren el archivo de configuración; por defecto =3000años, Fig. 2 - criterio config 11) - eliminar puntos de control duplicados en términos de edad y/o profundidad
- en varios casos, el punto de control de la parte superior del núcleo es de tipo
guess. El usuario puede especificar que el tipoguesssolo sea aceptado hasta cierta profundidad usandoguess_depth(por defecto es10cm, Fig. 2 - criterio config 12)
Además, el número y la distribución de estos puntos pueden indicar la incertidumbre temporal de las edades (Giesecke et al. 2014, Flantua et al. 2016). Un registro con pocos puntos en el periodo de interés tendrá mayor incertidumbre.
Porcentaje de carbono
Existen tres maneras de reportar fechas post-bomba de radiocarbono: 1) fechas modernas (<0 F14C años); 2) porcentaje de carbono moderno (pMC, normalizado a 100%); y 3) fracción de radiocarbono (F14C, normalizado a 1; Reimer et al. 2004). Actualmente no hay manera directa de saber si las fechas de Neotoma están en pMC o F14C. Si se detectan, el flujo exporta los registros sospechosos y el usuario debe especificar cuáles deben retrotransformarse (stop-check, Fig. 2). Para los seleccionados, se convierten los valores pMC a edades negativas con IntCal::pMC.age(), tras lo cual se usan las curvas post-bomba para calibrar a edades de calendario.
Aunque F14C se recomienda como estándar, en la práctica la mayoría se reporta como fechas normales de radiocarbono o como pMC. El flujo actualmente no gestiona F14C ya que no se han detectado casos. Si se encuentra con otras fuentes, se puede transformar usando el paquete {IntCal}.
02_Run_age_depth_models.R
Los modelos edad-profundidad individuales se estiman con el paquete {Bchron}, que estima la trayectoria probabilística bayesiana (modelo no paramétrico). Es apto para diversas combinaciones de tipos de puntos de control, outliers y reversals.
Si hay muchas edades a profundidades cercanas, pueden producirse problemas de inicialización y Bchron podría no converger. En ese caso, el grosor de los puntos duplicados se aumenta automáticamente por 0.01 (ver argumento artificialThickness en Bchron::Bchronology()).
Computación multinúcleo
Como crear modelos edad-profundidad para muchos registros puede ser intensivo, el flujo usa computación paralela. Detecta automáticamente los núcleos de la máquina (ajustable con number_of_cores en el archivo de configuración, Fig. 2 - criterio config 14). Los modelos se crean en lotes, con el tamaño determinado por el número de núcleos (batch size, Fig. 2 - criterio config 13; por defecto depende de number_of_cores). Si number_of_cores es 1, no usa el modo lote.
Si el cálculo del lote falla, el flujo omite ese lote y sigue con los demás. El usuario puede ajustar el tiempo de espera con el argumento time_per_record en RFossilpol::chron_recalibrate_ad_models().
Cada lote se intenta tres veces (ajustable con batch_attempts). Si se detiene el modelado, se usan los lotes exitosos previamente guardados. Si algún registro causa que R se congele completamente, su ID se guarda automáticamente en el archivo Crash (/Data/Input/Chronology_setting/Bchron_crash/) y se omite en futuras ejecuciones. Se recomienda revisar ese dataset en detalle.
Iteraciones
En el archivo de configuración, el número de iteraciones es 50,000 por defecto, descartando las primeras 10,000 (burn-in) y guardando cada iteración subsiguiente con un salto de 40 (thinning) (Fig. 2 - criterios config 15, 16, 17). El usuario puede cambiar esto con iteration_multiplier (Fig. 2 - criterio config 18). Así, se obtienen 1000 ((50k - 10k) / 40) valores posteriores. El número por defecto es robusto pero se puede aumentar (aunque incrementará el tiempo de espera).
Hay que tener en cuenta que modelar cientos de registros con muchas iteraciones es intensivo y puede llevar decenas de horas o días.
03_Predict_ages.R
Con un modelo exitoso, se estiman las edades de cada nivel. El flujo calculará la edad y la estimación de error (que engloba el 95% de los valores posteriores: límite superior e inferior). Como {Bchron} es probabilístico, es posible obtener posibles edades. Para ello, se extraen varios valores (por defecto = 1000), se calculan cuartiles, usando el cuantil 50 como edad final y los cuantiles 2.5 y 97.5 como límites. Todo se guarda como matriz de incertidumbre de edad (age_uncertainty). Todo el proceso es automático.
04_Save_AD_figures.R
La representación visual de todos los modelos edad-profundidad se guarda como PDFs en /Outputs/Figures/Chronologies/, divididos por región. Las propiedades se pueden cambiar en el archivo de configuración (image_width, image_height, image_units, image_dpi, text_size, line_size). Se recomienda revisarlas para buscar errores grandes, hiatos o extrapolaciones extremas.
05_Merge_chron_output.R
Las edades predichas se enlazan con todos los registros de las diferentes fuentes (Secciones I-III). Los niveles de cada registro se ordenan por edades predichas y el mismo orden se aplica a las tablas de conteos de polen.
V. Armonización: /05_Harmonisation/
Run_01_05.R- ejecuta todos los scripts dentro de esta carpeta01_Harmonisation.R- prepara todas las tablas de armonización y armoniza los conteos brutos.
Nota sobre armonización
El objetivo de la armonización taxonómica es estandarizar todos los nombres a nivel de sitio a los mismos morfotipos de polen (conjunto de morfotipos de polen y esporas usados para todos los registros), y así reducir el efecto de la incertidumbre taxonómica y la complejidad nomenclatural (ver literatura relevante en el Apéndice 1 de Flantua et al. 2023). Para ello, se puede crear una tabla de armonización que agrupa los morfotipos en el mayor nivel taxonómic que es muy probable que la mayoría de los analistas de polen lo identifiquen.
01_Harmonisation.R
Primero, el flujo de trabajo revisa las regiones de armonización presentes en los datos, definidas por el shapefile (ver Entrada de datos y Sección III), y confirma que exista una tabla de armonización por región (un punto stop-check, Fig. 2). Si falta alguna tabla (o se ejecuta por primera vez), el flujo crea automáticamente una tabla por región, con todos los nombres crudos de taxones de todos los registros de esa región. Cabe destacar que, para simplificar, nos referimos aquí a “nombres de taxón”, pero la identificación suele realizarse a nivel de familia o género, y se denominan mejor morfotipos.
Cada tabla de armonización tiene dos columnas:
taxon_namees el nombre original del taxón (morfotipo) en formato computacional (snake_case)level 1, que se usa para agrupar varios taxones (morfotipos) en unidades armonizadas, específicas del proyecto.
Para enlazar los nombres con la visualización original de Neotoma, el usuario puede usar la taxa_reference_table (ver proceso de limpieza de nombres).
El usuario puede también definir qué taxones deben eliminarse totalmente durante la armonización (marcados como delete), en caso de errores taxonómicos o proxies no deseados (ej. esporas). Se pueden añadir columnas extra (ej. level_2) y especificar qué niveles incluir ajustando el argumento harm_name en la función RFossilpol::harmonise_all_regions().
Con estas tablas, cada conjunto se armoniza para que todos los taxones que pertenezcan al mismo taxón armonizado (morfotipo) se sumen en cada nivel; esto aplica tanto para datos de conteo como de porcentaje. El proceso incluye una verificación automática de éxito de la armonización, revisando el número total de granos antes y después de armonizar (puede desactivarse cambiando el argumento pollen_grain_test a FALSE en la función RFossilpol::harmonise_all_regions()).
VI. Filtrado de datos: /06_Main_filtering/
Run_01_06.R- ejecuta todos los scripts dentro de esta carpeta01_Level_filtering.R- filtra niveles y registros según los criterios definidos por el usuario en el archivo de configuración
Nota sobre el filtrado de datos
Para obtener una compilación comprensiva de múltiples registros de polen fósil, mejorar la calidad general y responder preguntas de investigación de forma fiable, recomendamos reducir aún más la selección filtrando niveles individuales y/o registros completos. Todos los criterios de filtrado pueden ser ajustados por el usuario en el archivo de configuración:
filter_by_pollen_sum(Fig. 2 - criterio config 20) - si esTRUE, el flujo utiliza la cantidad de granos de polen contados en cada nivel como indicador de calidad.filter_by_age_limit(Fig. 2 - criterio config 24) - si esTRUE, se filtran registros que no cubran el periodo de interés definido por el usuario.filter_by_extrapolation(Fig. 2 - criterio config 25) - si esTRUE, se filtran niveles según el número de años extrapolados respecto al último punto de control de cronología usado.filter_by_interest_region(Fig. 2 - criterio config 27) - si esTRUE, se filtran niveles mayores aend_of_interest_period.filter_by_number_of_levels(Fig. 2 - criterio config 28) - si esTRUE, se filtran registros según el número de niveles.
Además, se pueden activar dos opciones más:
01_Level_filtering.R
Suma de granos de polen
El número de granos contados en cada nivel es un índice de calidad. Para una representación fiable de la vegetación, se recomienda contar más de 300 granos (Moore et al., 1991), aunque pueden usarse otros criterios (por ejemplo, 150; ver Djamali & Cilleros, 2020). En regiones árticas puede bastar con c. 100, en sitios mediterráneos puede llegar a 1000 (Birks & Birks, 1980), pero la preferencia del analista es clave. Números bajos suelen deberse a limitaciones de tiempo, pero también a fenómenos de preservación. Dado que el poder inferencial estadístico es proporcional al tamaño de muestra, recomendamos definir un mínimo de granos en cada nivel. Luego, se puede aceptar un registro solo si X% (por defecto 50, Fig. 2 - criterio config 23) de los niveles cumple el número aceptable.
Criterios de edad
Según el enfoque temporal, solo un subconjunto de registros será de interés. Por tanto, registros que no cubran el periodo de interés serán filtrados si filter_by_age_limit == TRUE.
Extrapolación de edad
La extrapolación de edades a muestras más allá del último punto de control incrementa la incertidumbre. Recomendamos seleccionar una extrapolación máxima (ej. 5000 años) respecto al último punto de control; niveles más viejos serán filtrados si filter_by_extrapolation == TRUE.
Periodo de interés
Niveles fuera del periodo de interés (end_of_interest_period) serán filtrados si filter_by_interest_region == TRUE.
Número de niveles
El número total de niveles es importante para futuros análisis. Registros con pocos niveles aportan poco y pueden ser fuentes de valores atípicos. Se recomienda definir un número mínimo (min_n_levels, Fig. 2 - criterio config 29); se filtrará si filter_by_number_of_levels == TRUE.
VII. Salidas/Outputs: /07_Outputs/
Run_01_07.R- ejecuta todos los scripts dentro de esta carpeta01_Pollen_diagrams.R- guarda los diagramas de polen de todos los registros02_Save_assembly.R- guarda el ensamblado de datos con las variables seleccionadas03_Save_references.R- guarda referencias y metadatos
01_Pollen_diagrams.R
Los diagramas de polen para todos los registros se crean usando el paquete {rioja}. Los datos armonizados se transforman automáticamente en proporción relativa para graficar. Los diagramas se guardan en /Outputs/Figures/Pollen_diagrams/, divididos por Región.
La función RFossilpol::plot_all_pollen_diagrams() genera automáticamente un PDF de los diagramas, listos para imprimir. El eje Y es, por defecto, la edad de los niveles (puede cambiarse a profundidad con el argumento y_var). El máximo de taxones por página se ajusta con max_taxa (por defecto, 20). Además, la función omite taxones muy raros, definido por min_n_occur.
02_Save_assembly.R
Una compilación armonizada y estandarizada, lista para análisis, se produce y guarda en /Outputs/Data/ como tibble (rds). El usuario puede seleccionar qué columnas deben estar presentes en la compilación final estableciendo select_final_variables == TRUE en el archivo de configuración. El flujo pedirá al usuario en la consola de R si incluir cada variable.
03_Save_references.R
El flujo guarda todos los metadatos y la información de citación necesarios para crear la compilación de datos. Todos los resultados están en /Outputs/Meta_and_references/. La función RFossilpol::proc_save_references() lo hace automáticamente, pero el usuario puede especificar qué información guardar usando el argumento user_sel_variables. Por defecto incluye:
"meta_table"- tabla de metadatos (data_assembly_meta.csv) contiene por defecto (la lista puede alterarse ajustando variables finales porselect_final_variables):- lista de todos los registros en la compilación final
- ubicación geográfica
- ambiente de depósito
- región continental asignada
- región de armonización asignada
- número final de niveles
- número de puntos de control de cronología usados en el modelado edad-profundidad
- curvas de calibración asignadas
- límites de edad de cada registro por fuente (Neotoma u otra)
- DOI (de Neotoma)
"author_table"- tabla de referencias (authors_meta.csv) con información sobre los conjuntos de datos utilizados, el principal contribuyente y su contacto."affiliation_table"- si se provee afiliación con datos distintos de Neotoma, la tabla de afiliaciones (affiliation_table) enlaza autores y afiliaciones."graphical_summary"- PDF (graphical_summary*.pdf) con tres figuras usandoRFossilpol::plot_graphical_summary():- mapa de ubicación geográfica de los registros
- gráfico de bastones con el número de registros por grupo
- límites de edad de los registros (línea por registro)
"reproducibility_bundle"- un zip (reproducibility_bundle.zip) con el archivo de configuración, todas las tablas stop-check, y todos los shapefiles. La idea es aumentar la reproducibilidad; este zip puede compartirse al publicar un proyecto creado con el flujo FOSSILPOL, o durante revisión para obtener feedback detallado antes de la publicación.